Fonte: ScienceDirect Abstrato Os tomates são cultivados e consumidos em quase todos os países do…

Epialelos de ocorrência natural determinam o acúmulo de vitamina E em frutos de tomate
Fonte: Nature Communications
Abstrato
O teor de vitamina E (VTE) é uma característica nutricional de baixa herdabilidade para a qual os determinantes genéticos são pouco compreendidos. Aqui, nos concentramos em um loci de característica quantitativa de TEV de tomate previamente detectado (QTL; mQTL 9-2-6 ) e identificamos o gene causal como aquele que codifica uma 2-metil-6-fitilquinol metiltransferase (ou seja, VTE3 (1)) que catalisa uma das etapas finais da biossíntese de γ- e α-tocoferóis, que são as principais formas de TEV. Por abordagens genéticas reversas, análises de expressão, perfis de siRNA e ensaios de metilação de DNA, demonstramos que mQTL 9-2-6 é um QTL de expressão associado à metilação diferencial de um retrotransposon SINE localizado na região promotora de VTE3 (1). A metilação do DNA promotor pode ser revertida espontaneamente, levando a diferentes epialelos afetando a expressão de VTE3 (1) e o conteúdo de VTE em frutas. Estas descobertas indicam, portanto, que os epialelos que ocorrem naturalmente são responsáveis pela regulação de um QTL metabólico nutricionalmente importante e fornecem evidência direta de um papel para a epigenética na determinação de características agronômicas.
Introdução
Os determinantes genéticos da qualidade nutricional têm sido estudados há muito tempo em muitas culturas importantes 1 , 2 , 3 , 4 , 5 . No entanto, os mecanismos que ligam os poucos determinantes genéticos identificados aos fenótipos finais são em grande parte desconhecidos. Isto é verdade para os tococromanóis, um grupo de compostos produzidos exclusivamente em organismos fotossintéticos 6 , 7 e que inclui tocoferóis e tocotrienóis (denominados vitamina E-VTE-), que possuem alto valor nutricional na dieta humana. Os tocoferóis são as moléculas que apresentam maior atividade de TEV 8 . Esses compostos consistem em um grupo de cabeça polar de cromanol (derivado do homogentisato) e uma cadeia lateral prenil lipofílica, produtos das vias do chiquimato plastidial e do metileritritol fosfato, respectivamente. A primeira etapa para a síntese de tocoferol é catalisada pela homogentisado fitil transferase (VTE2, EC 2.5. 1.-), que condensa o fitil 2P e o homogentizado para produzir 2-metil-6-fitilquinol. A partir deste precursor, são sintetizadas as quatro formas que ocorrem naturalmente. Enquanto γ- e α-tocoferol são produzidos pelas atividades sequenciais de dimetil-fitilquinol metil transferase (VTE3, EC 2.1.1.-), tocoferol ciclase (VTE1, EC 5.3.-.-) e tocoferol C-metil transferase (VTE4 , CE 2.1.1.95); A síntese de δ e β-tocoferol não requer que a atividade VTE3 seja formada meramente pela ação das duas últimas enzimas ( Fig. 1a ) 9 . O papel dos tocoferóis como antioxidantes lipossolúveis está bem documentado para plantas e humanos e ocorre por dois mecanismos: (i) doação de um átomo de hidrogênio a um radical peroxil de ácido graxo poliinsaturado altamente reativo, evitando assim uma reação em cadeia de peroxidação lipídica e (ii ) extinguindo física ou quimicamente espécies de oxigênio singlete, que danificam muitas moléculas biológicas, incluindo proteínas, DNA, carboidratos e ácidos graxos poliinsaturados 10 . Dado que a fotossíntese é uma importante fonte de oxigênio singleto, este mecanismo de extinção desempenha um papel importante na regulação funcional do fotossistema II, com implicações importantes nas respostas fisiológicas a condições de alta luminosidade, baixa temperatura e estresse osmótico 11 .
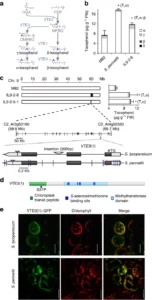
( a ) A via central do tocoferol nas plantas. Enzimas biossintéticas, homogentisado fitil transferase (VTE2), dimetil-fitilquinol metil transferase (VTE3), tocoferol ciclase (VTE1) e γ-tocoferol C-metil transferase (VTE4) estão em tipos azuis. Precursores e intermediários são: homogentisato (HGA), fitil 2P (PDP), metil-fitilquinol (MPBQ) e dimetil-fitilquinol (DMPBQ). ( b ) Composição e teor de tocoferol total (média ± SEM, n = 4) em frutos maduros de tomate das duas espécies parentais; S. lycopersicum (cv. M82) e S. pennellii (LA716), e a IL9-2-6 que mostra o QTL mapeado. ( c ) Mapeamento fino do QTL 9-2-6 para uma região centromérica do cromossomo 9 que abriga o alelo selvagem do gene VTE3 (1). Através da análise de 190 plantas F2 e 50 plantas F3, derivadas do cruzamento entre S. lycopersicum (cv. M82) e IL9-2-6, foi obtida uma sub-linhagem recombinante (IL9-2-6-1). O conteúdo de tocoferol (média ± SEM, n = 6) em frutos maduros de tomate destas linhas é mostrado no lado direito deste painel. A estrutura genômica da introgressão contida nesta linha é mostrada abaixo com setas pretas indicando genes transcricionalmente ativos. Uma visão ampliada dos alelos de S. lycopersicum e S. pennellii do gene VTE3 (1) mostra polimorfismos e inserções sinônimos (barras azuis) e não sinônimos (barra vermelha) no segundo íntron e na 3′UTR. As caixas pretas e cinza representam exons e 5′ e 3′ UTR, respectivamente. ( d ) Estrutura proteica da enzima VTE3 (1) do tomate indicando a substituição de aminoácidos (S37P). ( e ) A expressão transitória de ambos os alelos da proteína de fusão VTE3(1)::GFP em células mesofílicas de folhas de N. benthamiana indica direcionamento ao cloroplasto sob exame de microscopia confocal. Os asteriscos nos painéis b e c denotam diferenças estatisticamente significativas para o tocoferol total (T) e as formas de tocoferol indicadas ( teste t P< 0,05). O esquema do cromossomo 9 é baseado em distâncias físicas calculadas de acordo com a versão SL2.4 do genoma do tomate ( www.solgenomics.net ). A barra branca em e indica 25 μm.
A importância do tocoferol tanto para os organismos produtores quanto para os consumidores levou à pesquisa de loci de características quantitativas (QTL) que determinam o conteúdo de TEV 3 , 12 , 13 , 14 , 15 . No tomate, revelamos a existência de quatro QTL metabólicos (mQTL) que afetam os níveis totais de tocoferol nos frutos 9 . Estes quatro mQTL são mapeados nos cromossomos 6, 8 e 9. No entanto, dada a herdabilidade muito baixa (H 2 ) do conteúdo de TEV no tomate 16 , será difícil produzir frutos com teores elevados de TEV até que os determinantes genéticos e mecanismos subjacentes a esses QTL sejam totalmente compreendido.
Neste trabalho, relatamos o mapeamento fino do principal mQTL de TEV (mQTL 9-2-6 ) em tomate para um locus que codifica uma enzima VTE3. Mostramos que os alelos VTE3 (1) são expressos diferencialmente e que essas diferenças se correlacionam com diferenças na metilação do DNA das sequências promotoras e no acúmulo de RNA interferente curto (siRNAs) correspondentes. Consistente com o mQTL 9-2-6 sendo de natureza epigenética, descobrimos que a metilação do DNA no VTE3(1) varia consideravelmente entre as populações naturais de tomate e que o VTE3(1) pode reverter espontaneamente da forma metilada para a não metilada, com impacto no TEV conteúdo em frutas.
Resultados
Mapeamento fino de um importante tocoferol mQTL em frutos de tomate
Decidimos identificar o (s) gene (s) causal (is) abrigado (s) pelo mQTL específico da fruta para o conteúdo total e α-tocoferol que havia sido previamente mapeado no cromossomo 9 (mQTL 9-2-6 ) 9 ( Fig. 1b e Fig. Complementar .1a ). Para tanto, foi realizado mapeamento fino utilizando uma população F2 de 190 indivíduos, derivada de um cruzamento entre a linhagem parental S. lycopersicum (cv. M82) e a linhagem introgressiva 9-2-6 (IL9-2-6) ( abrangendo aproximadamente 3 Mb e contendo mais de aproximadamente 300 genes). Os marcadores moleculares C2_At3g63190 (58,9 Mb), VTE3(1)INDEL (59,6 Mb), C2_At4g02580 (60 Mb), 1531800 (61,0 Mb) e C2_At2g47890 (61,8 Mb) (Figura 1b suplementar) foram utilizados para triagem e o fragmento introgredido segregado 2:1:1 ( S.lyc / S.pen : S.lyc / S.lyc : S.pen / S.pen , χ2 = 0,98). Uma única linha recombinante entre os marcadores C2_At4g02580 e 1531800 foi identificada e autofecundada para dar origem à IL9-6-2-1 recombinante, que abriga apenas 769 Kb do fragmento introgresso original. A quantificação de TEV demonstrou que o mQTL de tocoferol total foi mantido nesta IL recombinante ( Fig. 1c ). Dos 56 supostos quadros de leitura abertos (ORFs) presentes neste intervalo de 789 Kb ( www.solgenomics.net ), 13 ORFs não são suportados por nenhum dado de expressão disponível (tags de sequência expressa ou leituras de RNA-seq) e apenas 36 mostram níveis de expressão detectáveis em frutos de tomate. Uma análise de anotação funcional do MapMan 17 revelou que destes 36, apenas um único gene ( locus VTE3 (1) -Solyc09g065730-), pertence à categoria Metabolismo Secundário ( Tabela Suplementar 1 ). Este gene apresentou alta similaridade (~ 85%) com uma dimetil-fitilquinol metil transferase de Arabidopsis (EC 2.1.1.-; Figura Complementar 1c, d ) que catalisa a conversão de 2-metil-6-fitilquinol em 2,3 -dimetil-5-fitilquinol, o precursor imediato do γ-tocoferol ( Fig. 1a ) 18 . Este gene está duplicado em várias espécies de eudicotiledôneas, incluindo S. lycopersicum ( Figura 1c Complementar ). O VTE3(1) do tomate possui 339 aminoácidos e contém os dois domínios funcionais característicos encontrados na proteína Arabidopsis , bem como um suposto peptídeo de trânsito do cloroplasto. Ao comparar a divergência alélica entre S. lycopersicum e S. pennelliiproteínas, foi encontrado um único polimorfismo de aminoácido localizado dentro de uma região pouco conservada do peptídeo de trânsito do cloroplasto ( Fig. 1d e Fig. Complementar 1d ). Ensaios de localização subcelular transitória realizados com alelos de S. lycopersicum e S. pennellii fundidos com GFP indicaram que ambos os alelos VTE3 (1) são direcionados para o cloroplasto onde ocorre a biossíntese de tocoferol ( Fig. 1e ). Tomados em conjunto, estes resultados sugerem que o TEV3 (1) é um dos principais determinantes do mQTL 9-2-6-1 para o conteúdo de tocoferol da fruta, mas é improvável que o(s) polimorfismo(s) causal(is) resida(m) na sequência de codificação.
Os níveis de mRNA de VTE3 (1) modulam o conteúdo de VTE em frutos de tomate
A fim de demonstrar o envolvimento do VTE3 (1) na determinação do teor de tocoferol nas frutas, silenciamos especificamente esse gene nas frutas por meio de um protocolo estabelecido de silenciamento gênico induzido por vírus, que se baseia na fluorescência da GFP como marcador visual 19 . Para este propósito, um fragmento de 380 pb da região codificadora de GFP foi fundido a um fragmento de 349 pb do gene de tomate VTE3 (1) para co-silenciamento. Após a infiltração dos pedúnculos inflorescentes, os setores silenciados por GFP dos frutos foram monitorados na planta ao longo do desenvolvimento e os tomates verdes maduros (MG) e maduros foram colhidos 42 e 60 dias após a infiltração, respectivamente. Os setores sem fluorescência foram dissecados ( Fig. 2a ) e os níveis de mRNA de VTE3 (1) foram medidos por PCR quantitativo (qPCR). Reduções de aproximadamente 60% foram observadas nos setores de pericarpo silenciados por GFP em relação aos frutos formados a partir de plantas infiltradas apenas com pTRV2-GFP ( Fig. 2b ). Em seguida, testamos o efeito do silenciamento do VTE3 (1) no metabolismo do tocoferol nas frutas. As formas de tocoferol dependentes de TEV3 (γ e α) apresentaram reduções significativas em ambos os estágios de desenvolvimento, enquanto as formas independentes de TEV3 (δ e β) aumentaram mais de duas vezes. Além disso, os níveis absolutos de α- e γ-tocoferol foram significativamente reduzidos em frutos maduros silenciados por VTE3 (1) ( Fig. 2c ). Estas alterações no metabolismo do tocoferol foram especificamente relacionadas ao silenciamento de VTE3 (1), uma vez que os níveis de mRNA dos outros genes da via biossintética do núcleo do tocoferol ( VTE1 , VTE3 (2) e VTE4 ) permaneceram inalterados neste experimento ( Figura 2a Complementar ). Além disso, uma análise do perfil metabólico dos tecidos silenciados não revelou alterações importantes, sugerindo que esta enzima não afeta o metabolismo primário. No entanto, aumentos de licopeno e α-caroteno foram detectados em frutos maduros nos quais o gene VTE3 (1) foi silenciado ( Figura 2b Complementar ) e essas alterações foram acompanhadas por um aumento da capacidade antioxidante ( Figura 2c Complementar ). Estes dados demonstram assim que a alteração dos níveis de ARNm de VTE3 (1) afecta directamente o conteúdo e a composição do tocoferol no tomate e, além disso, apoia hipóteses anteriores de que o estado redox do fruto é equilibrado por mecanismos que afectam as vias metabólicas associadas ao metabolismo do tocoferol e dos carotenóides 20 , 21 , 22 .
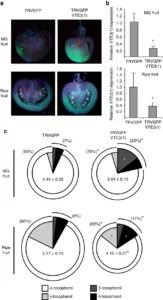
( a ) GFP (controle) e GFP – VTE3 (1) MG silenciado e frutos maduros colhidos de pedúnculos inflorescentes agroinfiltrados com silenciamento de genes induzidos por vírus de tomateiros que expressam GFP ( S. lycopersicum cv. Moneymaker). ( b ) Níveis relativos de mRNA de VTE3 (1) em GFP (controle) e GFP – VTE3 (1) frutos silenciados (média ± sem, n = 6). ( c ) Conteúdo de tocoferol (μg g −1 FW ± sem, n = 6), composição relativa de tocoferol e proporção de formas de tocoferol dependentes de VTE3 e independentes de VTE3 das mesmas frutas silenciadas mostradas em a são indicadas por números, gráficos de pizza e semicírculos, respectivamente. Observe que os tamanhos dos gráficos de pizza são proporcionais ao conteúdo médio total de tocoferol. Os asteriscos indicam diferenças estatisticamente significativas ( teste t P< 0,05).
A expressão específica dos alelos VTE3 (1) determina mQTL 9-2-6
Tendo demonstrado que a expressão de VTE3 (1) determina os níveis de tocoferol tanto no MG quanto nos estágios maduros, em seguida comparamos a expressão dos alelos cultivados e selvagens. De acordo com os perfis de conteúdo de tocoferol ( Fig. 1b ), o alelo selvagem apresentou níveis de mRNA significativamente mais elevados em frutos maduros de ambas as linhas de introgressão (IL9-2-6 e 9-2-6-1) em comparação com o controle parental receptor . Em outras palavras, QTL 9-2-6 é uma expressão QTL (eQTL; Fig. 3a ). O maior acúmulo de mRNA de VTE3 (1) após a introgressão do alelo selvagem no fundo de S. lycopersicum não foi restrito aos frutos maduros, mas também foi observado nas folhas e no desenvolvimento dos frutos ( Fig. 3b ). Além disso, estas diferenças na expressão de VTE3 (1) não são acompanhadas por alterações no nível de expressão de qualquer um dos outros genes que codificam as enzimas envolvidas na via biossintética do tocoferol ( Fig. 3c ). Além disso, a avaliação dos níveis de expressão de outras 19 ORFs presentes no segmento IL9-2-6-1 introgredido por qPCR mostrou resultados semelhantes aos depositados em bases de dados públicas (ted.bti.cornell.edu), exceto para três ORFs. No entanto, nenhuma destas três ORFs está relacionada com as vias metabólicas do TEV e a sua expressão não se correlaciona com os níveis de tocoferol no tomate. Além disso, os três ortólogos de A. thaliana não são coexpressos com nenhum gene envolvido na biossíntese de tocoferol 23 . Tomados em conjunto, estes resultados confirmam ainda que o VTE3(1) é o gene causal albergado pelo mQTL 9-2-6.
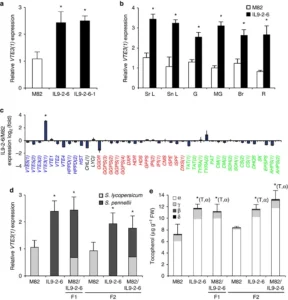
a ) Níveis de mRNA de VTE3 (1) em frutos maduros das linhagens parentais S. lycopersicum (cv. M82) e IL9-2-6 e a IL9-2-6-1 recombinante resultante (média ± sem, n = 5) . ( b ) Níveis de mRNA de VTE3 (1) nas folhas fonte (Sr L) e sumidouro (Sn L) e ao longo do desenvolvimento dos frutos (frutos verdes-G, verdes-MG maduros, breaker-Br e maduros-R) colhidos das linhagens parentais S. lycopersicum (cv. M82) e IL9-2-6 (média ± sem, n = 6). ( c ) níveis de mRNA quantificados em frutos maduros das linhagens parentais S. lycopersicum (cv. M82) e IL9-2-6 de todos os genes que codificam enzimas envolvidas no metabolismo do tocoferol do tomate de acordo com 24 (média ± sem, n = 3). Os genes pertencentes ao núcleo do tocoferol, associados às vias do metileritritol fosfato e do chiquimato são indicados em azul, preto, vermelho e verde, respectivamente. ( d , e ) mRNA de VTE3 (1), composição de tocoferol e níveis totais medidos em linhagens homozigóticas e heterozigóticas de VTE3 (1) para duas gerações (F1 e F2) derivadas de um cruzamento entre S. lycopersicum (cv. M82) e IL9- 2-6 (média ± sem, n =5). A contribuição de cada alelo VTE3 (1) ( S. lycopersicum e S. pennellii ) foi quantificada por PCR-transcriptase reversa quantitativa específica do alelo e indicada por barras cinza claro e escuro, respectivamente. Os asteriscos em a , b , c e d denotam diferenças estatisticamente significativas (teste de randomização P < 0,05 corrigido pela taxa de descoberta falsa em caso de testes múltiplos). Os asteriscos em e denotam diferenças estatisticamente significativas para o tocoferol total (T) e as formas de tocoferol indicadas ( teste t P< 0,05).
Para desvendar ainda mais a natureza do eQTL VTE3 (1), sua estabilidade transgeracional foi analisada em frutos maduros das linhagens híbridas F1 e segregantes F2 com as três possíveis combinações de alelos determinadas por um ensaio de expressão específico de alelo. No híbrido F1, o nível total de mRNA de VTE3 (1) é equivalente ao medido em IL9-2-6 como consequência da dominância do alelo selvagem. Da mesma forma, a expressão de VTE3 (1) apresenta o mesmo padrão nas linhas recombinantes F2 derivadas ( Fig. 3d ). Além disso, o conteúdo total de tocoferol segue o mesmo perfil dos níveis de mRNA de VTE3 (1) ( Fig. 3e ), sugerindo que o mQTL 9-2-6 e o eQTL de VTE3 (1) estão firmemente e estavelmente acoplados ao longo das gerações.
Revelando o mecanismo molecular do tocoferol QTL 9-2-6
Os níveis mais elevados de mRNA de VTE3 (1) em IL9-2-6 e IL9-2-6-1 podem ser o resultado de diferenças nas atividades do promotor transcricional entre os alelos domesticados e selvagens. Portanto, analisamos as regiões promotoras desses alelos por meio de uma busca de novo por elementos reguladores cis (CREs) 24 . O promotor do gene VTE3 (1) de S. lycopersicum contém sete CREs estatisticamente super-representados, enquanto o de S. pennelli contém quatro. Dois destes elementos são comuns às regiões promotoras VTE3 (1) de S. lycopersicum , S. pennellii e S. phureja ( Figura 3a suplementar ). Comparações adicionais entre as regiões promotoras de S. lycopersicum , S. pimpinellifolium , S. pennellii e S. phureja revelaram padrões distintos de inserções de elementos transponíveis (TE). Enquanto duas cópias da família Tc1-IS630-Pogo de transposons de DNA 25 são comuns entre todas as quatro espécies analisadas, indicando que estavam presentes no ancestral comum, um elemento retrotransposon da família SINE 26 é encontrado exclusivamente na espécie mais próxima, S. lycopersicum e S. pimpinellifolium , sugerindo uma inserção específica posterior na linhagem do tomate (seção Lycopersicon; Figura 3b suplementar ). Além disso, a região genômica que abrange este SINE é altamente metilada em frutos de S. lycopersicum (cv Ailsa Craig) ( //ted.bti.cornell.edu/epigenome/ ), principalmente em locais CHH, que é uma marca registrada do RNA metilação dirigida do DNA (RdDM) 27 . A fim de avaliar se a maquinaria RdDM pode estar envolvida no estabelecimento e manutenção da metilação do DNA nesta região, em seguida geramos perfis de siRNA-seq a partir de frutos maduros de S. lycopersicum , S. pennellii e IL9-2-6- 1. Descobrimos que um número considerável de espécies de 21-24 nt-siRNAs (sense e antisense) corresponde às sequências TE localizadas no promotor de S. lycopersicum ( Fig. 4a,b ), mas nenhuma em S. pennellii nem na IL9- 2-6-1. Por meio da digestão com enzima McrBC e PCR semiquantitativa específica de alelo, avaliamos o estado de metilação de três diferentes fragmentos do gene VTE3 (1): dois na região promotora (I e II) e um na sequência codificante (III). Os resultados revelaram que a região promotora proximal do alelo de S. lycopersicum que abriga o SINETE é hipermetilado, enquanto o selvagem, seja em seu próprio background ( S. pennellii ) ou no background M82 (IL9-2-6 e IL9-2-6-1), apresentou baixos níveis de metilação do DNA ( Fig. 4c ). Para melhor caracterizar esses epialelos, investigamos a metilação do DNA na resolução de um único nucleotídeo, utilizando tratamento com bissulfito do DNA genômico seguido de sequenciamento direcionado. Os resultados confirmaram a hipermetilação da região promotora (II) proximal de VTE3 (1) do alelo de S. lycopersicum nos três contextos genómicos ( Fig. 4d ). Como os siRNAs parecem estar envolvidos no estabelecimento do epialelo hipermetilado do TEV3 (1), testamos a existência de interações alélicas, como a paramutação 28 . Para determinar se este é o caso ou não, analisamos em seguida a metilação do DNA específico do alelo nos híbridos F1 e heterozigotos F2. Em consonância com os resultados da expressão ( Fig. 3d ), descobrimos que o status epigenético de cada alelo permanece inalterado nos híbridos, demonstrando que eles não são alvos de paramutação ( Fig. Complementar 4 ).
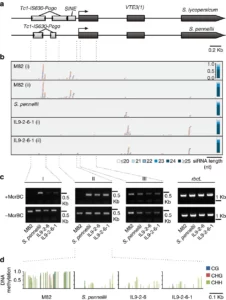
( a ) Gene VTE3 (1) e estrutura do promotor (janela de 4 Kb) mostrando exons (caixas pretas) e inserções de TE (caixas cinza claro). ( b ) Perfis de siRNA específicos de locus do VTE3(1) . As trilhas mostram a posição de mapeamento do siRNA único sentido (vermelho) e antisense (azul) obtido de frutos maduros de S. lycopersicum (cv. M82), S. pennellii (LA716) e IL9-2-6-1. A distribuição de comprimento dos siRNAs mapeados é representada à direita de cada trilha. siRNA de S. lycopersicum (cv. M82) e IL9-2-6-1 foram extraídos e analisados de dois conjuntos de frutos colhidos de plantas independentes (indicados por i e ii), enquanto de S. pennellii (LA716) foram extraídos e analisados a partir de um pool de cinco frutas. ( c ) Status de metilação do DNA avaliado por um tratamento de DNA com endonuclease sensível à metilação McrBC seguido de amplificação por PCR em duas regiões do promotor VTE3 (1) (I: distal e II: proximal), no primeiro exon do gene (III) e numa região codificadora do gene do cloroplasto Rubisco LS ( rbcL ) como controlo. O DNA foi extraído de pelo menos quatro frutos maduros dos genótipos S. lycopersicum (cv. M82), S. pennellii (LA716), IL9-2-6 e IL9-2-6-1. ( d ) Metilação de resíduos de citosina nos locais CG, CHG e CHH (linhas azul, vermelha e verde, respectivamente) testadas por sequenciamento de bissulfito da região promotora proximal (região II em c) em DNA extraído de frutos maduros de S. lycopersicum (cv. M82), S. pennellii (LA716), genótipos IL9-2-6 e IL9-2-6-1.
A fim de avaliar se a regulação dependente da metilação do alelo VTE3 (1) de S. lycopersicum é um mecanismo específico de M82 ou melhor, um mecanismo que pode ser estendido a outras origens genéticas, analisamos ainda a expressão de VTE3 (1) e DNA metilação em uma série alélica de raças locais andinas de tomate, três cultivares de referência comercial e uma espécie próxima ( S. pimpinellifolium -LA1589) e uma espécie selvagem distantemente relacionada ( S. habrochaites -LA407). Esses acessos foram coletados em diferentes localizações geográficas e testados nas mesmas condições de campo. Resultados publicados anteriormente demonstraram que eles diferem nos conteúdos de tocoferol e carotenóides 24 . Descobrimos que tanto a expressão de VTE3 (1) quanto a metilação de sua região promotora proximal variam entre os diferentes genótipos. Além disso, a abundância de mRNA de VTE3 (1) correlaciona-se inversamente com os níveis de metilação da região promotora proximal ( Fig. 5a e Fig. Complementar 5a ). Consequentemente, os cultivares com níveis mais elevados de mRNA de VTE3 (1) acumulam mais γ-tocoferol em frutos maduros ( Figura 5b Complementar ). A análise da sequência de um intervalo de 850 pb abrangendo o promotor do gene VTE3 (1) dos 13 genótipos aqui estudados revelou a presença de polimorfismos de nucleotídeo único e pequenos InDels, mas estes não se correlacionam com diferenças no nível de expressão ou metilação do DNA (Figs. Suplementares 5a e 6 ).

( a ) Níveis de mRNA de VTE3 (1) (média ± sem, n = 3) e status de metilação do DNA avaliado por um tratamento de DNA com endonuclease sensível à metilação McrBC, seguido por amplificação por PCR da região promotora proximal (analisada na Fig. 4d ) em frutos maduros de oito variedades crioulas de tomate andino ( S. lycopersicum ), três cultivares de referência comercial e as espécies silvestres S. pimpinellifolium (LA1589) e S. habrochaites (LA407). ( b ) Perfis de siRNAs específicos de locus de VTE3(1) . As trilhas mostram a posição de mapeamento de siRNA exclusivo de sentido (vermelho) e antisense (azul) de frutos maduros colhidos de S. lycopersicum (cv. M82) cultivado em casa de vegetação e de um ensaio de campo realizado em Mendoza (Argentina, 32° 50′S, 68° 52′W e 900 msnm). A distribuição de comprimento dos siRNAs mapeados é representada em uma caixa à direita de cada trilha. O gráfico de barras no painel direito mostra o número normalizado (± sem, n = 2) de siRNAs mapeados na região promotora proximal de VTE3(1) (indicado por uma linha azul abaixo do painel de trilhas). Os siRNAs foram extraídos e analisados de dois conjuntos de frutos colhidos de plantas independentes, cada um (indicado por i e ii) ( c ) Metilação de resíduos de citosina nos locais CG, CHG e CHH (linhas azul, vermelha e verde, respectivamente) testada por bissulfito direcionado sequenciação da região promotora proximal (analisada na Fig. 4d ) a partir de ADN extraído de um conjunto das mesmas amostras que em b . O gráfico de barras no painel direito mostra a quantificação da metilação do DNA para cada contexto. ( d , e ) Composição de tocoferol e níveis de mRNA de VTE3 (1) (média ± sem, n = 3) das mesmas amostras de b e c . ( e ) Atividade transcricional de promotores de alelos selvagens e cultivados medidos por um ensaio transitório utilizando o vetor pGreenII 0800-LUC conforme descrito nos Métodos (± sem, n ≥6). O asterisco no painel b , c e e denota diferenças estatisticamente significativas ( teste t , teste de Fisher e teste de randomização, respectivamente; P < 0,05). O asterisco em d denota diferenças estatisticamente significativas para o tocoferol total (T) e as formas de tocoferol indicadas ( teste t P< 0,05).



